Grippe aviaire : Huit génotypes de virus H5N1 trouvés en France
L’épizootie 2021-2022 d’influenza aviaire a été marquée par une plus forte diversité génétique des virus influenza aviaire H5N1, un ayant été majoritaire dans le sud-ouest et un autre majoritaire en Pays de La Loire.
L’épizootie 2021-2022 d’influenza aviaire a été marquée par une plus forte diversité génétique des virus influenza aviaire H5N1, un ayant été majoritaire dans le sud-ouest et un autre majoritaire en Pays de La Loire.
Les analyses phylogénétiques de 149 échantillons de virus influenza aviaire hautement pathogène (IA HP) du sous-type H5N1, réalisées par le laboratoire national de référence sur l’influenza aviaire (LNR), installé à l’Anses de Ploufragan (22), mettent en évidence huit génotypes de virus appartenant tous au sous-type H5N1.
Selon Béatrice Grasland, responsable du LNR, « les virus IA HP d’un génotype n’ont pas muté pour donner un autre génotype. Il s’agit de virus réassortants qui ne présentent pas tous les mêmes gènes. »
Pour rappel, le matériel génétique d’un virus IA est composé de huit brins. Un virus est dit « réassortant » quand il est issu de la rencontre de deux virus IA différents au sein d’un même hôte, qui donne naissance à un nouveau virus composé d’un mélange de segments issus des deux virus « parents ».
Une variabilité génétique observée en Europe
Selon les experts du LNR, cette diversité est le signe de nombreuses introductions sur le territoire via l’avifaune sauvage, contrairement à l’épizootie précédente avec moins d’introductions et avec la prédominance du sous type H5N8.
La même tendance est observée à l’échelle européenne, avec au moins vingt nouveaux génotypes H5 identifiés. Ils résulteraient de multiples « réassortiments » des virus hautement pathogènes (IA HP) entre eux et avec des virus autochtones faiblement pathogènes (IA FP).
Sur ces vingt génotypes européens, dix-neuf concernent les virus H5N1 et un génotype le virus H5N2 IAHP.
En 2020/2021, cinq sous types H5 avaient été détectés (H5N8, H5N1, H5N5, H5N3 et H5N4) dont un seul génotype pour H5N1.

Au moins cinq introductions dans le Sud-ouest
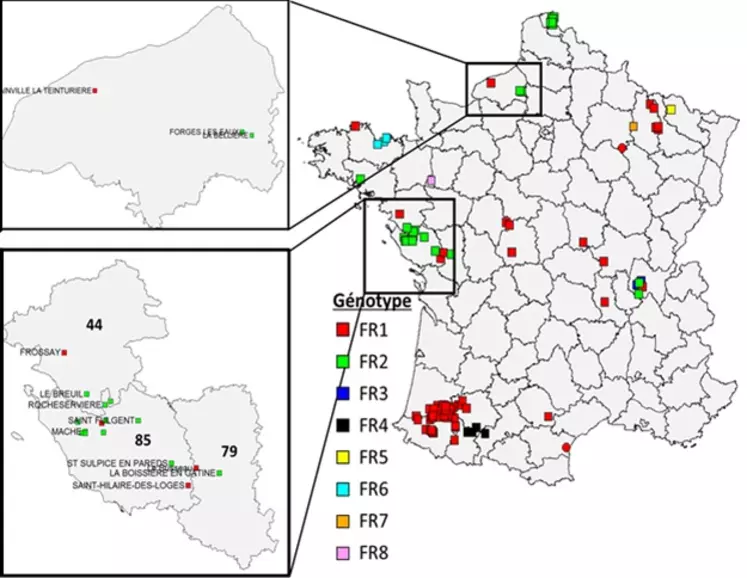
Le premier génotype - dénommé FR1 - a été détecté chez des oiseaux sauvages sur une grande partie du territoire tout au long de l’hiver 2021-2022. Il a également été identifié majoritairement dans les élevages du sud-ouest (Gers, Pyrénées-Atlantiques, Landes et Hautes-Pyrénées, mais aussi en Vendée début janvier et dans l’Indre. FR1 a aussi été retrouvé en Italie, République tchèque, Pologne, Russie, Estonie et Angleterre.
Une analyse génétique plus fine suggère qu’au moins cinq introductions de FR1 ont eu lieu dans les élevages du sud-ouest : une dans le Gers, une seconde sur la commune de H (1) dans les Landes (40) et de C en Pyrénées atlantiques (64), une troisième dans les communes d’O (64) et de S (40) puis dans le Tarn (liens épidémiologiques directs prouvés entre élevages), une quatrième à D, P et A (64) ainsi qu’à B (40) (liens directs) et enfin une cinquième introduction dans le sud-est des Landes qui est ensuite devenue majoritaire. C’est ce virus qui a diffusé vers l’est, le nord-ouest et le sud-est des Landes, puis atteint le Gers, les Pyrénées-Atlantiques et les Hautes-Pyrénées.
(1) Le laboratoire national de référence ne souhaite pas que leur nom apparaisse.
Une seule étincelle en Vendée
Le génotype FR2 a touché des élevages de volailles (Nord, Seine-Maritime, Vendée), une basse-cour (Morbihan) et des oiseaux sauvages (Ain). Le génotype a aussi été identifié en Russie, aux Pays-Bas, en Belgique et en Croatie.
En Vendée, l’étroite parenté des différents génomes viraux détectés à partir de fin janvier 2022 indiquent un lien direct ou très proche entre ces virus FR2. Ce qui suggère une transmission qui s’est généralisée de proche en proche, par aéroportage des plumes et des poussières contaminées et/ou par contacts indirects (matériel, transport, paille, personnel…). En revanche, il n’est pas possible de trancher entre l’hypothèse d’une introduction par l’avifaune migratrice remontante et celle d’une avifaune autochtone dans la mesure où ce génotype a préalablement été détecté en France.
Interrogée sur le lien entre génotype et pathogénicité, Béatrice Grasland répond qu’elle « n’a pas d’indications qu’il y ait une différence de virulence entre des virus FR1 et des virus FR2. »
Six autres génotypes mineurs dans les élevages
Le génotype FR3 a été uniquement détecté chez des oiseaux sauvages de l’Ain et en Italie. Le génotype FR4 est très localisé et spécifique. Il a été détecté trois fois sur une oie cendrée sauvage dans les Hautes-Pyrénées fin décembre, dans une basse-cour de Haute-Garonne environ 10 jours plus tard, mi-février dans un élevage des Hautes-Pyrénées. Aucune séquence européenne apparentée n’est actuellement décrite dans les banques de données. Les génotypes FR5, FR6, FR7 et FR8 ont été détectés sur des oiseaux sauvages (Moselle, Côtes-d’Armor, Marne) et dans un élevage de Mayenne.
Le Laboratoire national de référence poursuit ses investigations sur un plus grand nombre d’échantillons, notamment sur la zone Dordogne-Lot dernièrement contaminée et pour laquelle les analyses phylogénétiques ne sont pas terminées.











